Strukturní biologie signálních proteinů
Na této straně najdete

O oddělení
Naše skupina se zabývá strukturní biologií (vztahy mezi strukturou a funkcí určitých skupin bílkovin), konkrétně se zaměřujeme na proteiny, které se účastní přenosu signálu v buňce. Mezi metody, které využíváme, patří příprava proteinů a jejich komplexů, základní biofyzikální charakterizace, charakterizace mezimolekulových interakcí a studium struktury a interakčních povrchů. Všechny tyto metody nám umožňují lépe pochopit detaily, jak je regulována aktivita a funkce protein-proteinových komplexů. Zabýváme se především výzkumem v těchto oblastech:
- Strukturní biologie 14-3-3 proteinů a jejich vazebných partnerů
- Studium inhibice ubiquitin ligásy Nedd4-2 pomocí proteinu 14-3-3
- Charakterizace interakcí mezi transkripčním faktorem FOXO4 a tumorovým supresorem p53
- Mechanismus regulace proteinkinasy ASK1 pomocí TRX a proteinu 14-3-3
- Mechanismus regulace proteázy kaspazy-2 pomocí proteinu 14-3-3
Projekty
Úspěchy
Cena Otto Wichterle
Prémie Otto Wichterle pro Dalibora Koška.
Dalibor Košek obdržel prémii Otto Wichterle pro vynikající mladé vědecké pracovníky AV ČR v roce 2022.
NOVÉ POZNATKY O STRUKTUŘE KOMPLEXU FOXO4:p53 – KLÍČOVÉHO FAKTORU V REGULACI STÁRNUTÍ BUNĚK (9.4.2022)
Transkripční faktor p53 brání tomu, aby se vlivem stresu staly z normálních buněk buňky nádorové. Za stresových podmínek p53 interaguje s dalším transkripčním faktorem FOXO4 (Forkhead box O 4) a společně pak zvyšují produkci proteinu p21, což spouští proces stárnutí buňky (senescenci). Molekulární mechanismus zvyšování transkripce p21 je však stále nejasný. V práci publikované v časopise Protein Science vědecké týmy Dr. Obšilové (FGÚ AV ČR), prof. Obšila (PřF UK a FGÚ AV ČR) a jejich kolegů z ÚOCHB AV ČR charakterizovali vzájemné interakce proteinů p53 a FOXO4 na molekulární úrovni. Nové poznatky o struktuře komplexu naleznou využití při vývoji specifických inhibitorů interakce mezi oběma proteiny, a následně pak při vývoji nových léčiv zaměřených na selektivní eliminaci senescentních buněk.
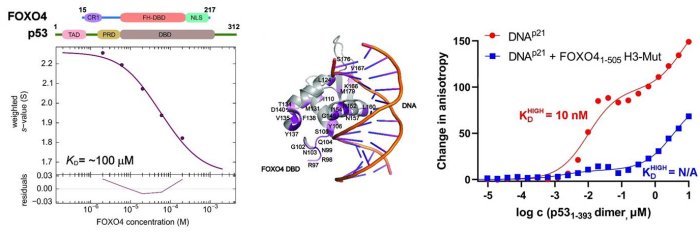
V této strukturní studii vědci provedli podrobnou charakterizaci interakcí v komplexu FOXO4:p53 pomocí integrovaného přístupu zahrnujícího analytickou ultracentrifugaci, nukleární magnetickou rezonanci a chemické zesítění spojené s hmotnostní spektrometrií. Protože FOXO4 i p53 mají více domén (viz. obr), studovali roli jednotlivých domén a neuspořádaných segmentů obou proteinů a zmapovali jejich interakční rozhraní. Zjistili, že zcela zásadní pro celkovou stabilitu komplexu p53:FOXO4 je interakce mezi transaktivační doménou TAD p53 a Forkhead doménou FOXO4. Na tvorbě komplexu se dále podílejí kontakty zahrnující N-koncový neuspořádaný segment FOXO4, C-koncovou negativní regulační doménu p53 a DNA-vazebné domény obou proteinů. Měřením vazby na DNA dále zjistili, že tvorba komplexu p53:FOXO4 blokuje vazbu p53 na DNA, aniž by však ovlivnila DNA-vazebné vlastnosti FOXO4.
Vlevo, analýza interakce mezi FOXO4 a p53 pomocí analytické ultracentrifugace, metodou sedimentační rychlosti. Uprostřed, změny v chemických posunech 15N-značeného FOXO4 v přítomnosti p53 získané z 1H-15N HSQC spekter namapované na krystalovou strukturu komplexu FOXO4 DBD:DNA. Vpravo, měření změn anisotropie fluorescence ukazující, že tvorba komplexu inhibuje vazbu p53 na DNA.
Mandal R, Kohoutova K, Petrvalska O, Horvath M, Srb P, Veverka V, Obsilova V and Obsil T. FOXO4 interacts with p53 TAD and CRD and inhibits its binding to DNA. Protein Sci. roč. 31, č. 5 (2022), č. článku e4287. IF = 6.725. DOI: 10.1002/pro.4287.
Naše nové dva články v Communications Biology
Two accepted papers in Communications Biology: July and August 2021
Pavel Pohl, Rohit Joshi, Olivia Petrvalska, Tomas Obsil and Veronika Obsilova
Commun. Biol. 2021 July 22; 4(1):899.
Matej Horvath, Olivia Petrvalska, Petr Herman, Tomas Obsil and Veronika Obsilova
Commun. Biol. 2021 August 19; 4(1):986.
IF = 6.268
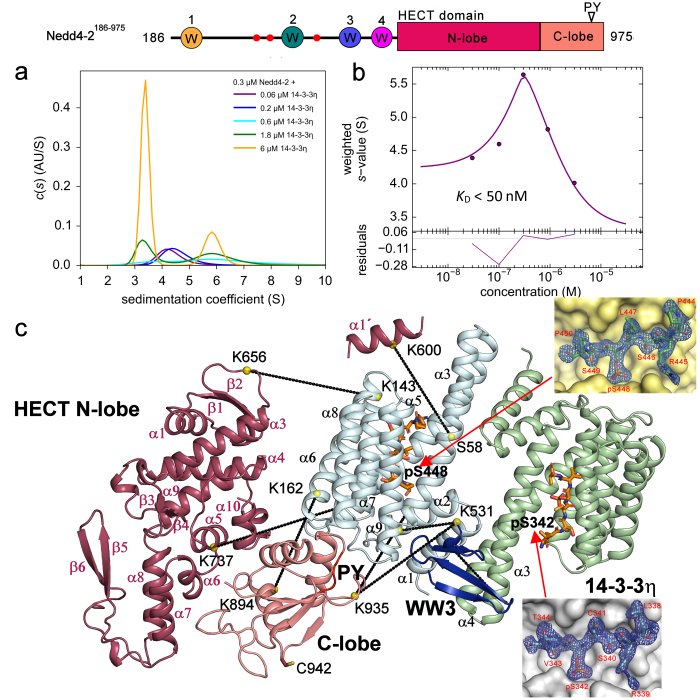
Pohl et al. investigated the structural basis of Nedd4-2 regulation by 14-3-3 and found that phosphorylated Ser342 and Ser448 are the main residues that facilitate 14-3-3 binding to Nedd4-2. The authors propose that the Nedd4-2:14-3-3 complex then stimulates a structural rearrangement of Nedd4-2 through inhibiting interaction of its structured domains.
Characterization of the interaction between Nedd4-2 and 14-3-3 in solution. a) Sedimentation coefficient distributions (c(s)) , b) Isotherm of weight-averaged sedimentation coefficients (sw) derived from SV-AUC analysis, c) SAXS-based structural model of the complex with the crystal structures of the Nedd4-2 phosphopeptides (Pohl et al. 2021).
Horvath et al. structurally and biochemically characterized the full-lenght human DAPK-14-3-3 complex to investigate the effects of binding to DAPK2 on its dimerization, activation by dephosphorylation of Ser318, and Ca2+/calmodulin binding. Their results provide mechanistic insights into 14-3-3-mediated DAPK2 inhibition and highlight the potential of the DAPK2:14-3-3 complex as a target for anti-inflammatory therapies.
Inovativní metoda regulace Forkhead transkripčního faktoru FOXO3
Studie otištěná v prestižním časopise eLife identifikuje nízkomolekulární sloučeniny, které interagují s transkripčním faktorem Forkhead box O3 (FOXO3) a modulují tak jeho aktivitu.
Protein FOXO3 s charakteristickou DNA-vazebnou doménou je součástí podtřídy O rodiny Forkhead transkripčních faktorů. Tyto transkripční faktory jsou v savčích buňkách klíčovými regulátory buněčné homeostázy, diferenciace, délky života a řízené buněčné smrti. Aktivita FOXO3 také významně přispívá k rezistenci rakovinných buněk vůči chemo- a radioterapii. Nejnovější studie rovněž ukázaly, že se DNA-vazebná doména (DBD) FOXO proteinů účastní protein-proteinových interakcí s dalšími klíčovými regulátory délky života, buněčné smrti a lékové rezistence. Reverzibilní inhibice aktivity FOXO3 prostřednictvím nízkomolekulárních sloučenin by tak mohla podpořit protinádorové imunitní odpovědi.
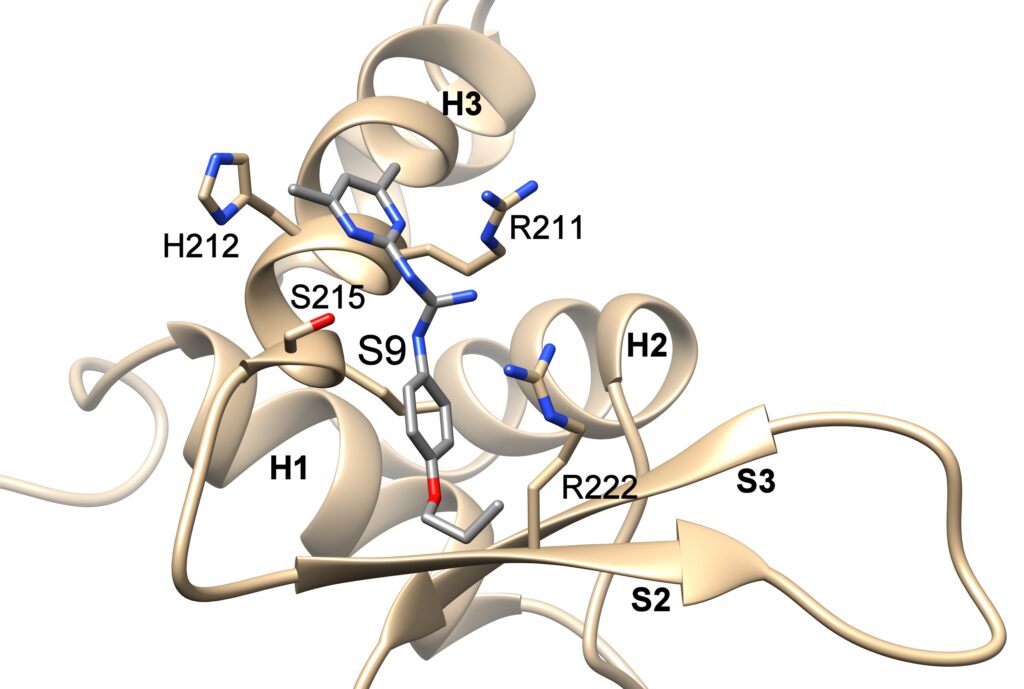
K inhibici aktivity FOXO3 bylo nejdříve nezbytné identifikovat nízkomolekulární sloučeniny, které by blokovaly interakci mezi FOXO3 DBD a DNA. Na základě struktury FOXO3 DBD a FOXO4 DBD bylo vytvořeno šest různých farmakoforových modelů, které byly využity pro in silico screening databází nízkomolekulárních sloučenin. U vybraných sloučenin byla následně testována schopnost inhibovat funkci FOXO3 jak in vitro, tak i v nádorových buněčných kulturách. Interakce sloučenin s FOXO3 DBD byly charakterizovány pomocí metod NMR spektroskopie a molekulového dokování.
Týmy dr. Veroniky Obšilové z Fyziologického ústavu AV ČR z BIOCEV a prof. Tomáše Obšila z Přírodovědecké fakulty UK ve spolupráci se skupinou prof. M. J. Ausserlechnera z Medical University Innsbruck, Rakousko identifikovaly sloučeniny S9 a její oxalátovou sůl S9OX jako látky schopné inhibovat FOXO3 v nádorových buňkách. Vědci zároveň vyslovili domněnku, že díky svému způsobu vazby na FOXO3 DBD mohou také S9 sloučeniny bránit protein-proteinovým interakcím mezi FOXO3 a jeho vazebnými partnery. Výhodou těchto látek je, že nejsou imunogenní, což umožňuje nastavení dávky a délky aplikace tak, aby byla posílena protinádorová imunita a zároveň byly omezeny nežádoucí efekty inhibice FOXO proteinů u kmenových buněk a somatických tkání. Další výzkum bude zaměřen na zjištění, jestli lze S9 využít jako základ pro vývoj sloučenin schopných specificky modulovat funkce jednotlivých FOXO transkripčních faktorů.
Původní studie: Hagenbuchner J*, Obsilova V*, Kaserer T*, Kaiser N, Rass B, Psenakova K, Docekal V, Alblova M, Kohoutova K, Schuster D, Aneichyk T, Vesely J, Obexer P, Obsil T+, Ausserlechner MJ+ Modulating FOXO3 transcriptional activity by small, DBD-binding molecules. eLife. 2019, 8(Dec 4), e48876. doi: 10.7554/eLife.48876. IF = 7.551
* Equall contribution.+ Corresponding authors.
Sloučeniny S9 blokují DNA-vazebný povrch Forkhead transkripčního faktoru FOXO3. Na obrázku je znázorněn strukturní model DNA-vazebné domény FOXO3 s navázanou sloučeninou S9 založený na datech z měření jaderné magnetické resonance a molekulového dokování.
Nejlepší práce vydaná ve Physiological Research s mladými vědeckými pracovníky v roce 2018
We obtained the first prize for the best publication in Physiological Research with the young authors from for the year 2018.
Nejlepší publikace s autory z FGÚ za rok 2017
We got the prize for the best publication of the Institute of Physiology for year 2017 for the publication:
Alblova M, Smidova A, Docekal V, Vesely J, Herman P, Obsilova V, Obsil T. :
Molecular basis of the 14-3-3 protein-dependent activation of yeast neutral trehalase Nth1.
Proc Natl Acad Sci U S A. (2017) 114:E9811-E9820. IF = 9.504.
Náš nový článek v PNAS: struktura komplexu proteinu 14-3-3 s enzymem Nth1.
Our new paper in PNAS:
Alblova M, Smidova A, Docekal V, Vesely J, Herman P, Obsilova V, Obsil T.
Molecular basis of the 14-3-3 protein-dependent activation of yeast neutral trehalase Nth1.
Proc Natl Acad Sci U S A. 2017 Oct 30. pii: 201714491. doi: 10.1073/pnas.1714491114.
https://www.ncbi.nlm.nih.gov/pubmed/29087344
The 14-3-3 proteins, a family of highly conserved scaffolding proteins ubiquitously expressed in all eukaryotic cells, interact with and regulate the function of several hundreds of partner proteins. Yeast neutral trehalases (Nth), enzymes responsible for the hydrolysis of trehalose to glucose, compared with trehalases from other organisms, possess distinct structure and regulation involving phosphorylation at multiple sites followed by binding to the 14-3-3 protein. Here we report the crystal structures of yeast Nth1 and its complex with Bmh1 (yeast 14-3-3 isoform), which, together with mutational and fluorescence studies, indicate that the binding of Nth1 by 14-3-3 triggers Nth1’s activity by enabling the proper 3D configuration of Nth1’s catalytic and calcium-binding domains relative to each other, thus stabilizing the flexible part of the active site required for catalysis. The presented structure of the Bmh1:Nth1 complex highlights the ability of 14-3-3 to modulate the structure of a multidomain binding partner and to function as an allosteric effector. Furthermore, comparison of the Bmh1:Nth1 complex structure with those of 14-3-3:serotonin N-acetyltransferase and 14-3-3:heat shock protein beta-6 complexes revealed similarities in the 3D structures of bound partner proteins, suggesting the highly conserved nature of 14-3-3 affects the structures of many client proteins.
Článek v JBC: strukturní náhled na inhibici proteinkinasy ASK1 prostřednictvím proteinu 14-3-3
Our new paper in JBC: August 2016 Structural Insight into the 14-3-3 Protein-Dependent Inhibition of Protein Kinase ASK1.
Petrvalska O, Kosek D, Kukacka Z, Tosner Z, Man P, Vecer J, Herman P, Obsilova V and Obsil T. J. Biol. Chem. jbc.M116.724310.
First Published on August 11, 2016, doi:10.1074/jbc.M116.724310
Apoptosis signal-regulating kinase 1 (ASK1, also known as MAP3K5), a member of the mitogen-activated protein kinase kinase kinase (MAP3K) family, regulates diverse physiological processes. The activity of ASK1 is triggered by various stress stimuli and is involved in the pathogenesis of cancer, neurodegeneration, inflammation, and diabetes. ASK1 forms a high molecular mass complex whose activity is, under non-stress conditions, suppressed through interaction with thioredoxin and the scaffolding protein 14-3-3. The 14-3-3 protein binds to the phosphorylated S966 motif downstream of the ASK1 kinase domain. The role of 14-3-3 in the inhibition of ASK1 has yet to be elucidated. In this study, we performed structural analysis of the complex between the ASK1 kinase domain phosphorylated at S966 (pASK1-CD) and the 14-3-3ζ protein. Small Angle X-ray Scattering (SAXS) measurements and chemical cross-linking revealed that the pASK1-CD:14-3-3ζ complex is dynamic and conformationally heterogeneous. In addition, structural analysis coupled with results of phosphorus NMR and time-resolved tryptophan fluorescence measurements suggest that 14-3-3ζ interacts with the kinase domain of ASK1 in close proximity to its active site, thus indicating this interaction might block its accessibility and/or affect its conformation.
Our new paper in JBC: Role of the EF-hand-like Motif in the 14-3-3 Protein-mediated Activation of Yeast Neutral Trehalase Nth1.
Kopecka M, Kosek D, Kukacka Z, Rezabkova L, Man P, Novak P, Obsil T, Obsilova V.
J Biol Chem. 2014 May 16;289(20):13948-61.
http://www.ncbi.nlm.nih.gov/pubmed/24713696
Trehalases hydrolyze the non-reducing disaccharide trehalose amassed by cells as a universal protectant and storage carbohydrate. Recently, it has been shown that the activity of neutral trehalase Nth1 from Saccharomyces cerevisiae is mediated by the 14-3-3 protein binding that modulates the structure of both the catalytic domain and the region containing the EF-hand-like motif, whose role in the activation of Nth1 is unclear. In this work, the structure of the Nth1·14-3-3 complex and the importance of the EF-hand-like motif were investigated using site-directed mutagenesis, hydrogen/deuterium exchange coupled to mass spectrometry, chemical cross-linking, and small angle x-ray scattering. The low resolution structural views of Nth1 alone and the Nth1·14-3-3 complex show that the 14-3-3 protein binding induces a significant structural rearrangement of the whole Nth1 molecule. The EF-hand-like motif-containing region forms a separate domain that interacts with both the 14-3-3 protein and the catalytic trehalase domain. The structural integrity of the EF-hand like motif is essential for the 14-3-3 protein-mediated activation of Nth1, and calcium binding, although not required for the activation, facilitates this process by affecting its structure. Our data suggest that the EF-hand like motif-containing domain functions as the intermediary through which the 14-3-3 protein modulates the function of the catalytic domain of Nth1.
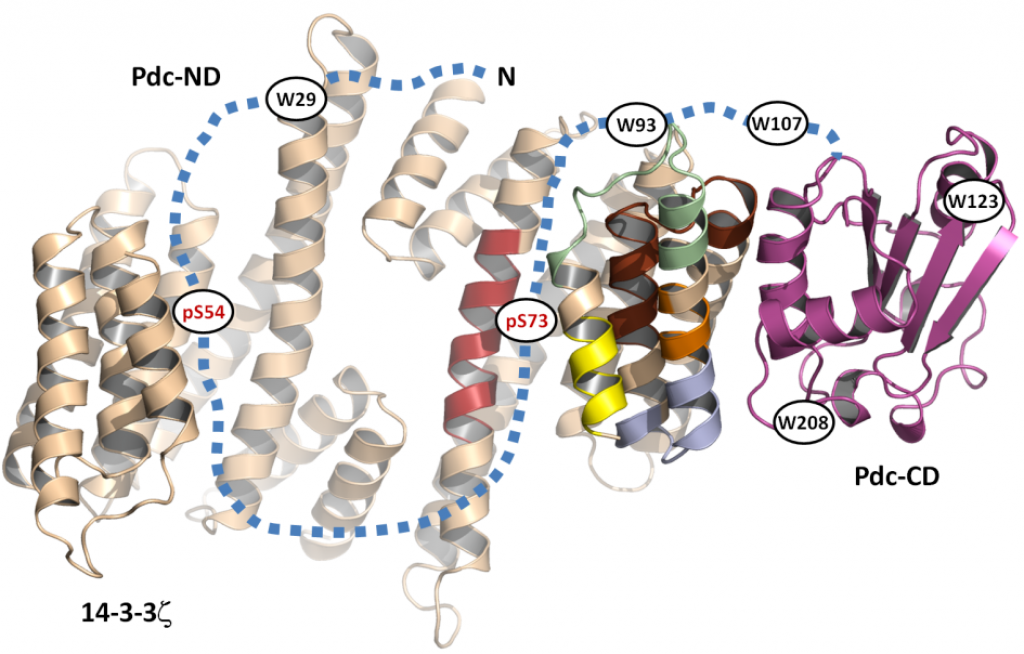
Our New Paper in JBC: Structural Characterization of Phosducin and its Complex with the 14-3-3 Protein
Kacirova M, Kosek D, Kadek A, Man P, Vecer J, Herman P, Obsilova V and Obsil T
J. Biol. Chem. jbc.M115.636563. First Published on May 13, 2015, doi:10.1074/jbc.M115.636563
Kacirova M, Kosek D, Kadek A, Man P, Vecer J, Herman P, Obsilova V and Obsil T
http://www.ncbi.nlm.nih.gov/pubmed/25971962
Phosducin (Pdc), a highly conserved phosphoprotein involved in the regulation of retinal phototransduction cascade, transcriptional control, and modulation of blood pressure, is controlled in the phosphorylation-dependent manner including the binding to the 14-3-3 protein. However, the molecular mechanism of this regulation is largely unknown. Here, the solution structure of Pdc and its interaction with the 14-3-3 protein were investigated using small angle X-ray scattering, time-resolved fluorescence spectroscopy and hydrogen-deuterium exchange coupled to mass spectrometry. The 14-3-3 protein dimer interacts with Pdc using surfaces both inside and outside its central channel. The N-terminal domain of Pdc, where both phosphorylation sites and the 14-3-3 binding motifs are located, is intrinsically disordered protein which reduces its flexibility in several regions without undergoing dramatic disorder-to-order transition upon binding to 14-3-3. Our data also indicate that the C-terminal domain of Pdc interacts with the outside surface of the 14-3-3 dimer through region involved in Gtβγ binding. In conclusion, we show that the 14-3-3 protein interacts with and sterically occludes both the N- and C-terminal Gtβγ binding interfaces of phosphorylated Pdc, thus providing a mechanistic explanation for the 14-3-3-dependent inhibition of Pdc function.
Tomáš Obšil byl jmenován profesorem
Prof. RNDr. Tomáš Obšil, Ph.D. byl jmenován profesorem na Přírodovědecké fakultě UK, obor Fyzikální chemie. Profesorská promoce proběhla 18.12.2014.
Hlávkova medaile 2014
16.12.2014: Nadace Josefa, Marie a Zdeňka Hlávky ocenila Mgr. Hanu Janouškovou jako nejlepší postgraduální studentku 2. lékařské fakulty, Karlovy University v Praze.
Our new paper in JBC: Biophysical and Structural Characterization of the Thioredoxin-binding Domain of Protein Kinase ASK1 and Its Interaction with Reduced Thioredoxin.
Kosek D, Kylarova S, Psenakova K, Rezabkova L, Herman P, Vecer J, Obsilova V, Obsil T.
J Biol Chem. 2014 Aug 29;289(35):24463-74.
http://www.ncbi.nlm.nih.gov/pubmed/25037217
Apoptosis signal-regulating kinase 1 (ASK1), a mitogen-activated protein kinase kinase kinase, plays a key role in the pathogenesis of multiple diseases. Its activity is regulated by thioredoxin (TRX1) but the precise mechanism of this regulation is unclear due to the lack of structural data. Here, we performed biophysical and structural characterization of the TRX1-binding domain of ASK1 (ASK1-TBD) and its complex with reduced TRX1. ASK1-TBD is a monomeric and rigid domain that forms a stable complex with reduced TRX1 with 1:1 molar stoichiometry. The binding interaction does not involve the formation of intermolecular disulfide bonds. Residues from the catalytic WCGPC motif of TRX1 are essential for complex stability with Trp(31) being directly involved in the binding interaction as suggested by time-resolved fluorescence. Small-angle x-ray scattering data reveal a compact and slightly asymmetric shape of ASK1-TBD and suggest reduced TRX1 interacts with this domain through the large binding interface without inducing any dramatic conformational change.
Získání grantu GA ČR
14-10061S – Mechanismus regulace kinasové aktivity proteinkinasy ASK1 (řešitelé: T. Obšil, PřF UK, a V. Obšilová, FgÚ AV ČR, v.v.i.)
Studentský Velemlok pro nejlepšího pedagoga
21. 2. 2014 – doc. RNDr. Tomáš Obšil oceněn studenty PřF UK jako nejlepší vyučující v oboru chemie
Cenu Studentský Velemlok pro nejlepšího pedagoga v roce 2013 převzal docent Tomáš Obšil z rukou děkana Přírodovědecké fakulty Univerzity Karlovy v Praze 21. 2. 2014. Pedagogové jsou na toto ocenění navrhováni studenty na základě výsledků hodnocení jejich výuky ve studentské anketě.
Publikace
Kumar; J. P. - Košek; Dalibor - Durell; S. R. - Miller Jenkins; L. M. - Debnath; S. - Coussens; N. P. - Hall; M. D. - Appella; D. H. - Dyda; F. - Mazur; S. J. - Appella; E. Crystal structure and mechanistic studies of the PPM1D serine/threonine phosphatase catalytic domain. Journal of Biological Chemistry. 2024; 300(8); 107561.
IF = 4.0
Masaryk; Jakub - Kale; Deepika - Pohl; Pavel - Ruiz-Castilla; F. J. - Zimmermannová; Olga - Obšilová; Veronika - Ramos; J. - Sychrová; Hana The second intracellular loop of the yeast Trk1 potassium transporter is involved in regulation of activity; and interaction with 14–3-3 proteins. . 2023; 21(April); 2705-2716.
IF = 4.4